Setiap organisme hidup, dari bakteri bersel tunggal hingga manusia yang kompleks, bergantung pada satu molekul utama untuk menyimpan instruksi genetiknya: Asam Deoksiribonukleat, atau DNA. Molekul heliks ganda ini adalah cetak biru kehidupan. Namun, agar kehidupan dapat berlanjut, berkembang biak, dan tumbuh, cetak biru ini harus disalin dengan sempurna. Proses penyalinan ini, yang dikenal sebagai replikasi DNA, adalah salah satu proses molekuler paling fundamental dan menakjubkan di dalam sel.
Replikasi DNA terjadi selama fase spesifik dari siklus sel yang disebut fase S (Sintesis), tepat sebelum sel membelah (baik melalui mitosis maupun meiosis). Tujuannya adalah untuk menghasilkan dua molekul DNA anak yang identik secara genetik dari satu molekul DNA induk. Proses ini bersifat semikonservatif, sebuah konsep yang dibuktikan oleh Meselson dan Stahl. Ini berarti bahwa setiap molekul DNA baru yang terbentuk terdiri dari satu untai “lama” (dari molekul induk) dan satu untai “baru” yang baru disintesis.
Untuk mencapai prestasi ini, sel mengerahkan serangkaian enzim dan protein yang bekerja bersama seperti mesin yang terkoordinasi dengan presisi tinggi. Proses ini dapat dibagi menjadi tiga tahap utama: inisiasi (permulaan), elongasi (pemanjangan), dan terminasi (pengakhiran).
1. Inisiasi: Mempersiapkan Garpu Replikasi
Replikasi tidak dimulai secara acak di sepanjang kromosom. Ia dimulai di lokasi spesifik yang disebut Titik Asal Replikasi (Origins of Replication – Ori). Pada prokariota seperti bakteri, biasanya hanya ada satu Ori pada kromosom sirkuler mereka. Sebaliknya, eukariota, dengan genom mereka yang jauh lebih besar dan linear, memiliki ribuan Ori di setiap kromosom, memungkinkan replikasi terjadi secara bersamaan di banyak tempat untuk menghemat waktu.
Proses inisiasi dimulai ketika sekelompok protein inisiator mengenali dan mengikat urutan DNA spesifik di Ori. Pengikatan ini menyebabkan DNA sedikit terbuka. Ini kemudian merekrut enzim kunci pertama, seperti yang ditunjukkan pada gambar pertama Anda: Helikase.
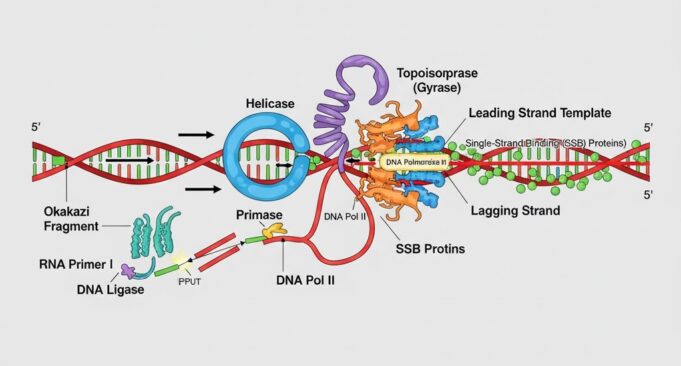
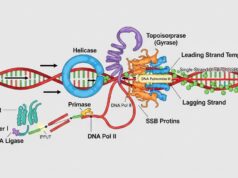
- DNA Helikase: Enzim ini berfungsi seperti “pembuka ritsleting”. Ia menggunakan energi dari hidrolisis ATP untuk secara aktif memutus ikatan hidrogen yang menyatukan pasangan basa (Adenin dengan Timin, Guanin dengan Sitosin). Saat Helikase bergerak maju, ia membuka pilinan heliks ganda DNA, menciptakan dua untai tunggal yang terpisah. Struktur berbentuk Y yang terbentuk di mana DNA dibuka ini disebut garpu replikasi (replication fork).
- Enzim Girase (Topoisomerase): Seperti yang diilustrasikan dalam diagram alur pertama, pembukaan heliks oleh Helikase menciptakan masalah. Bayangkan memuntir tali yang terdiri dari dua untaian; saat Anda membuka satu ujung, ujung lainnya akan menjadi semakin terpilin (ini disebut supercoiling atau tegangan torsi). Jika dibiarkan, ketegangan ini akan menghentikan replikasi. Di sinilah Enzim Girase (jenis dari Topoisomerase II) berperan. Ia bekerja di depan garpu replikasi, secara cerdik memotong sementara salah satu atau kedua untai DNA, membiarkannya berotasi untuk melepaskan tegangan, dan kemudian menyambungkannya kembali.
- Protein Pelindung Untas Tunggal (PPUT / SSB): Setelah dipisahkan oleh Helikase, untai-untai DNA tunggal yang kini terbuka menjadi rentan. Basa-basa hidrofobik mereka yang terekspos secara alami ingin berpasangan kembali satu sama lain (membentuk heliks ganda lagi) atau dilipat menjadi struktur jepit rambut yang kompleks. Untuk mencegah hal ini dan melindungi DNA dari kerusakan oleh enzim perusak (nuklease), Protein Pelindung Untas Tunggal (PPUT), atau Single-Strand Binding proteins (SSB), segera menempel dan melapisi untai-untai tunggal tersebut. Mereka menstabilkan DNA dalam keadaan terpisah, menjaganya tetap lurus dan tersedia sebagai cetakan untuk langkah berikutnya.
2. Elongasi: Membangun Untai Baru
Setelah garpu replikasi stabil dan cetakan DNA terbuka, mesin sintesis yang sebenarnya mulai bekerja. Pemain utamanya adalah keluarga enzim yang disebut DNA Polimerase. Namun, enzim ini memiliki dua keterbatasan penting:
- Mereka hanya dapat menambahkan nukleotida baru ke ujung 3′ (ujung hidroksil -OH) dari untai yang sedang tumbuh. Akibatnya, sintesis DNA selalu berlangsung dalam arah 5′ ke 3′.
- Mereka tidak dapat memulai sintesis “dari nol” (de novo). Mereka membutuhkan titik awal—sebuah untai pendek yang sudah ada untuk ditempeli, yang disebut primer.
Untuk mengatasi keterbatasan kedua, enzim lain yang disebut Primase masuk. Primase adalah jenis RNA Polimerase yang dapat memulai dari nol. Ia mensintesis segmen pendek RNA (sekitar 5-10 nukleotida) yang komplementer dengan cetakan DNA. Primer RNA ini menyediakan ujung 3′-OH yang dibutuhkan oleh DNA Polimerase untuk memulai tugasnya.
Karena kedua untai DNA dalam heliks ganda bersifat antiparalel (satu berjalan 5′-ke-3′ dan pasangannya 3′-ke-5′), sintesis pada kedua cetakan yang terpisah itu terjadi dengan cara yang sangat berbeda.
- Sintesis Untai Pengawal (Leading Strand): Satu untai cetakan, yang disebut leading strand template, berjalan dalam arah 3′-ke-5′ relatif terhadap pergerakan garpu replikasi. Ini adalah skenario ideal bagi DNA Polimerase (khususnya DNA Polimerase III pada prokariota). Setelah satu primer RNA dibuat oleh Primase di titik Ori, DNA Polimerase III dapat menempel dan terus menambahkan nukleotida DNA secara kontinu. Ia “mengikuti” tepat di belakang Helikase saat heliks terbuka, mensintesis untai baru dalam satu potongan panjang yang tidak terputus menuju garpu replikasi.
- Sintesis Untai Teralawal (Lagging Strand) dan Fragmen Okazaki: Untai cetakan lainnya, lagging strand template, menghadirkan tantangan besar. Ia berjalan dalam arah 5′-ke-3′. Karena DNA Polimerase harus mensintesis dalam arah 5′-ke-3′ (artinya ia harus “membaca” cetakan dalam arah 3′-ke-5′), ia harus bergerak menjauhi garpu replikasi, berlawanan dengan arah pembukaan heliks.
Ini diselesaikan melalui proses yang cerdik namun diskontinu (terputus-putus), seperti yang diilustrasikan pada gambar kedua Anda:
- Saat Helikase membuka segmen DNA baru, Primase membuat primer RNA baru di dekat garpu replikasi.
- DNA Polimerase III menempel pada primer RNA ini dan mensintesis segmen pendek DNA, bergerak menjauhi garpu, sampai ia menabrak primer RNA dari segmen sebelumnya.
- Segmen pendek DNA-RNA hibrida ini disebut Fragmen Okazaki.
- DNA Polimerase III kemudian melepaskan diri dan melompat ke primer RNA baru yang baru saja dibuat “di hulu” (lebih dekat ke garpu replikasi).
- Proses ini berulang-ulang, menciptakan serangkaian Fragmen Okazaki yang terputus-putus di sepanjang untai lagging.
Setelah ini, ada proses “pembersihan” yang krusial. Primer RNA tidak boleh ada di dalam molekul DNA akhir. Di sinilah enzim lain, DNA Polimerase I, berperan. Enzim ini memiliki aktivitas eksonuklease 5′-ke-3′, yang memungkinkannya “mengunyah” dan menghilangkan primer RNA di depannya, sambil secara bersamaan menggunakan aktivitas polimerase 5′-ke-3′ untuk mengisi celah yang ditinggalkan dengan nukleotida DNA yang benar.
Akhirnya, setelah semua primer RNA diganti dengan DNA, masih ada “celah” (nick) kecil di tulang punggung gula-fosfat antara Fragmen Okazaki yang berdekatan. Enzim terakhir, DNA Ligase, bekerja seperti “lem molekuler”. Ia menggunakan ATP untuk menciptakan ikatan fosfodiester terakhir, menyegel celah tersebut dan menyatukan semua Fragmen Okazaki menjadi satu untai DNA yang utuh dan kontinu.
3. Fidelitas dan Terminasi: Memastikan Akurasi dan Penyelesaian
Akurasi (Fidelitas): DNA Polimerase adalah mesin yang sangat akurat. Namun, dengan kecepatan sintesis ribuan basa per detik, kesalahan (misalnya, memasangkan A dengan C) terkadang terjadi. Untuk mengatasinya, DNA Polimerase III memiliki fungsi proofreading (koreksi) bawaan. Ia memiliki aktivitas eksonuklease 3′-ke-5′. Jika ia secara tidak sengaja menambahkan basa yang salah, ia dapat “mundur” satu langkah, memotong basa yang salah tersebut, dan kemudian mencoba lagi dengan basa yang benar. Mekanisme koreksi ini secara drastis mengurangi tingkat kesalahan replikasi, memastikan stabilitas genetik.
Terminasi (Pengakhiran): Proses ini akhirnya harus berhenti.
- Pada prokariota dengan kromosom sirkuler, dua garpu replikasi bergerak berlawanan arah dari Ori dan akhirnya bertemu di sisi lain lingkaran. Protein khusus mengikat di situs terminasi ini, menghentikan Helikase dan melepaskan seluruh kompleks replikasi.
- Pada eukariota dengan kromosom linear, terminasi lebih kompleks dan menimbulkan “Masalah Replikasi Ujung” (End Replication Problem). Ketika primer RNA terakhir di ujung untai lagging (di ujung kromosom) dihilangkan, tidak ada ujung 3′-OH bagi DNA Polimerase I untuk mengisinya. Akibatnya, setiap siklus replikasi akan meninggalkan celah kecil, menyebabkan kromosom memendek sedikit demi sedikit setiap kali sel membelah. Untuk melindungi gen-gen penting dari penghapusan, ujung kromosom eukariotik memiliki tutup pelindung yang disebut telomer (urutan DNA berulang yang tidak mengkode apa pun). Enzim khusus bernama telomerase (aktif di sel punca dan sel germinal) dapat memperpanjang telomer ini, tetapi di sebagian besar sel somatik, telomer terus memendek, yang diyakini berkontribusi pada proses penuaan sel.
Replikasi DNA adalah simfoni molekuler yang rumit. Dari pembukaan heliks oleh Helikase dan stabilisasi oleh PPUT/SSB, hingga pelepasan tegangan oleh Girase, dan sintesis yang kontras antara untai leading yang kontinu dan untai lagging yang terfragmentasi (Fragmen Okazaki), setiap langkah diatur dengan sempurna. Proses ini memastikan bahwa setiap sel anak menerima salinan cetak biru kehidupan yang lengkap dan akurat, memungkinkan pertumbuhan, perbaikan, dan pewarisan sifat dari generasi ke generasi.
REPLIKASI DNA — Infografik Interaktif
Klik kartu di bawah untuk membuka/menutup. Tahap: Inisiasi → Elongasi → Terminasi + kontrol kualitas (proofreading).
Origin of Replication (Ori)
Urutan DNA tempat replikasi dimulai
- Prokariota: biasanya 1 Ori (kromosom sirkuler).
- Eukariota: banyak Ori per kromosom (linear).
- Protein inisiator mengenali Ori dan membuka DNA.
Helikase
“Pembuka ritsleting” DNA
Memutus ikatan H antar basa menggunakan ATP → membentuk garpu replikasi.
SSB
Protein pelindung untai tunggal
Melapisi ssDNA agar tidak re-anneal/struktur jepit rambut dan mencegah degradasi.
Topoisomerase (Girase)
Penghilang lilitan (supercoiling)
Potong–putar–sambung DNA untuk meredakan tegangan di depan garpu.
Primase (RNA pol.)
Meletakkan primer RNA
Memberi ujung 3′-OH agar polimerase dapat mulai menambah dNTP.
DNA Polimerase III
Enzim utama sintesis 5′→3′
- Leading: kontinu mengikuti helikase.
- Lagging: diskontinu → Fragmen Okazaki.
Fragmen Okazaki
Potongan pendek pada untai lagging
Setiap fragmen dimulai dari primer baru; kemudian primer diganti DNA.
DNA Ligase
“Lem” fosfodiester
Menyegel nick antar fragmen sehingga tulang punggung DNA utuh.
Proofreading 3′→5′
Koreksi kesalahan oleh polimerase
Eksonuklease menghapus basa salah → fidelitas tinggi.
Terminasi Prokariota
Dua garpu bertemu di situs ter
Protein terminasi menghentikan helikase dan melepaskan replisom.
Telomer & Telomerase
Khusus kromosom linear eukariota
Primer terakhir di ujung lagging dihapus → pemendekan. Telomerase memperpanjang ulangan telomer (aktif di sel punca/germinal).
Aturan Arah
Selalu disintesis 5′→3′
Artinya polimerase membaca cetakan 3′→5′; ini menjelaskan kenapa untai lagging butuh banyak primer.
Inti Pesan
Replikasi DNA adalah kolaborasi enzimatik presisi: Helikase membuka, SSB menstabilkan, Topoisomerase mengurangi lilitan, Primase memberi start, Polimerase menyusun, Ligase menyegel, dan Proofreading menjamin akurasi; pada eukariota, Telomerase menjaga ujung kromosom.
Latihan Replikasi DNA — 15 Soal (ID/EN)
Klik jawaban, lalu tekan Lihat Jawaban / Show Answer pada setiap soal. (Tidak ada skor total — fokus ke pemahaman.)